本中心提供多種生物資訊分析方法,包含分子對接計算(Molecular Docking)、分子模擬(Molecular Modeling)、電腦虛擬篩選(Virtual Screening)及定量結構活性關係計算(QSAR)等服務技術,並搭配國家高速網路與計算中心提供之軟硬體設備,進行相關模擬計算。
技術簡介
一、 分子對接計算(Molecular Docking)
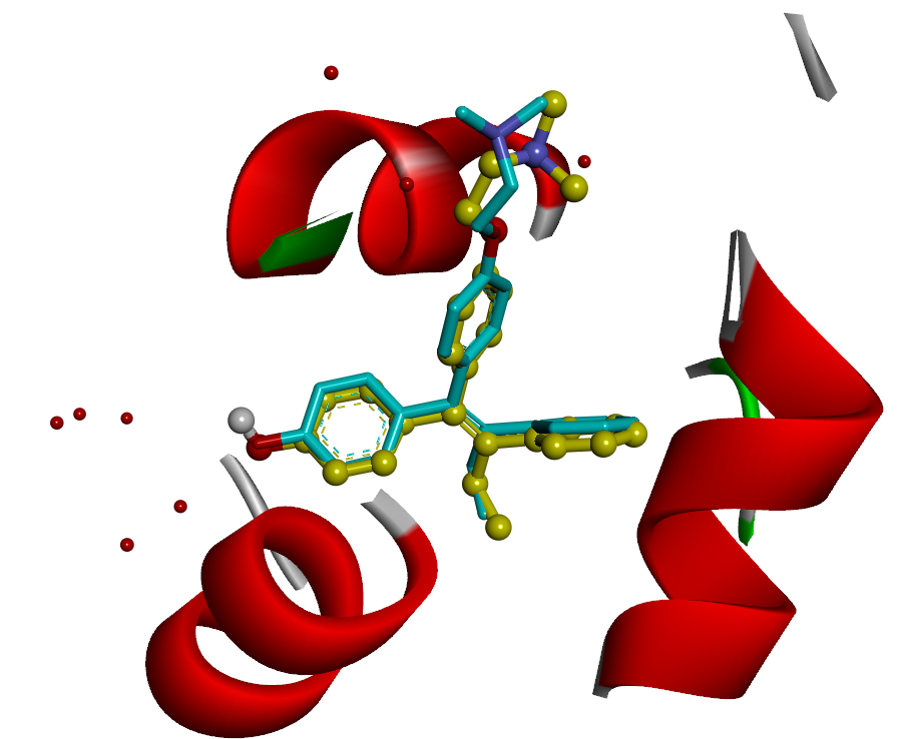
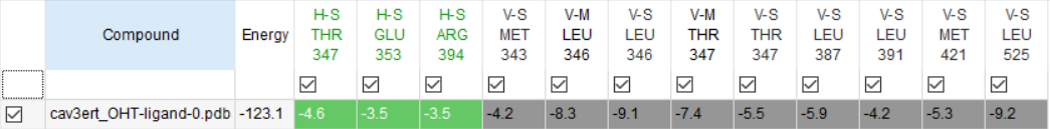
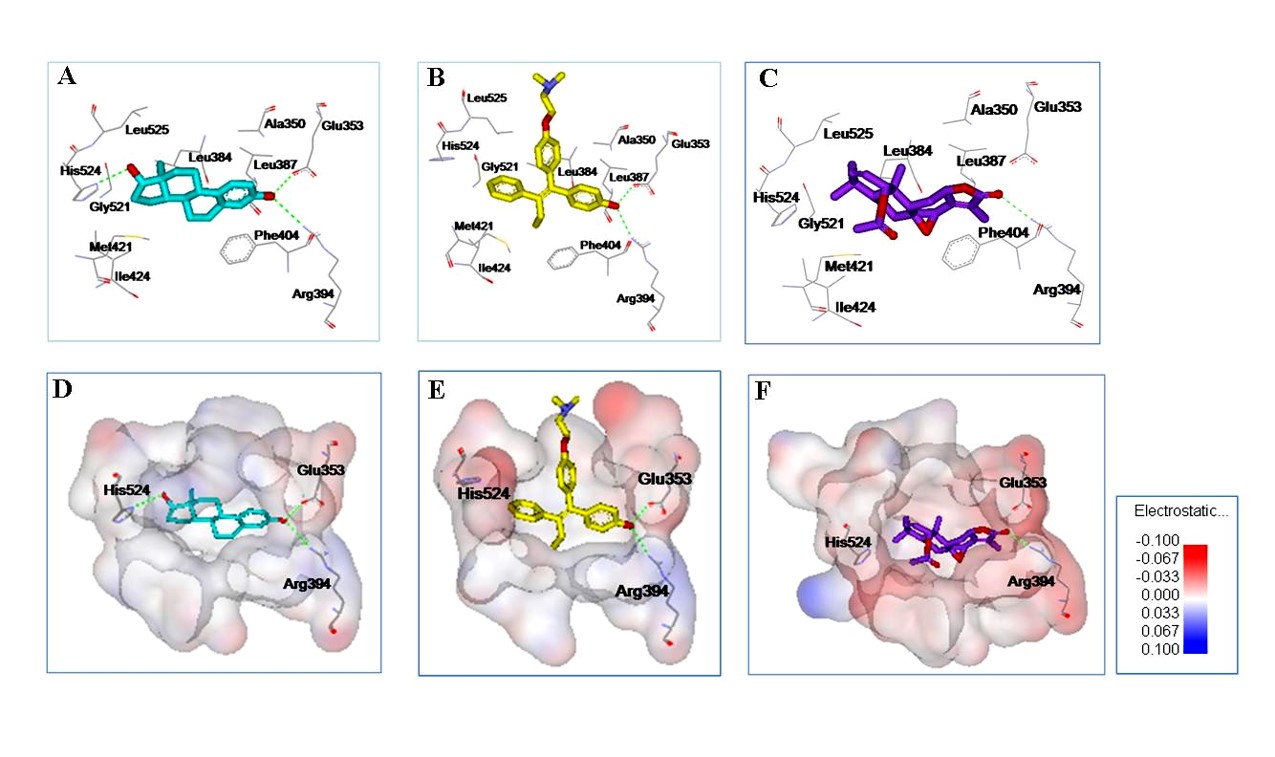
本中心可運用多種小分子對接計算軟體,例如:AutoDock、iGEMDOCK、GOLD等軟體進行小分子與蛋白質活化位之間的對接計算,搭配基因演算法或拉馬克基因演算法作為搜尋化合物形態的方法,並以分子模擬方法(Molecular Modeling)進一步了解其相互之間的交互作用力大小,作用力關係及其作用的距離。
分子對接計算可提供新藥開發研究者進一步的分子生物資訊,包含小分子或蛋白質的氫鍵作用力提供及被提供者數目、小分子與蛋白質活化位的極性與非極性作用關係、小分子在蛋白質活化位的構形表現等資訊,提供以結構為基礎之小分子藥物優化設計方向。
二、 電腦虛擬篩選(Virtual Screening)
本中心可運用多種小分子對接計算軟體,藉由對接計算方法針對特定蛋白質的活化位進行電腦虛擬篩選,從小分子巨量資料庫(例如ZINC Database)中針對特定蛋白質的活化位篩選出有影響潛力的小分子。
電腦虛擬篩選技術具有高速且低廉的優勢,並利用計算模擬方法做為化合物開發及修飾的參考資訊,具有處理大規模資料之優勢,可幫助生物醫學研究者,可針對特定蛋白結構找出有潛力的小分子,也可進行後續小分子抗癌或抗發炎等生物活性實驗,若從生物活性驗證中找到具活性之小分子,亦可進一步規劃接續之訊息傳遞路徑實驗。
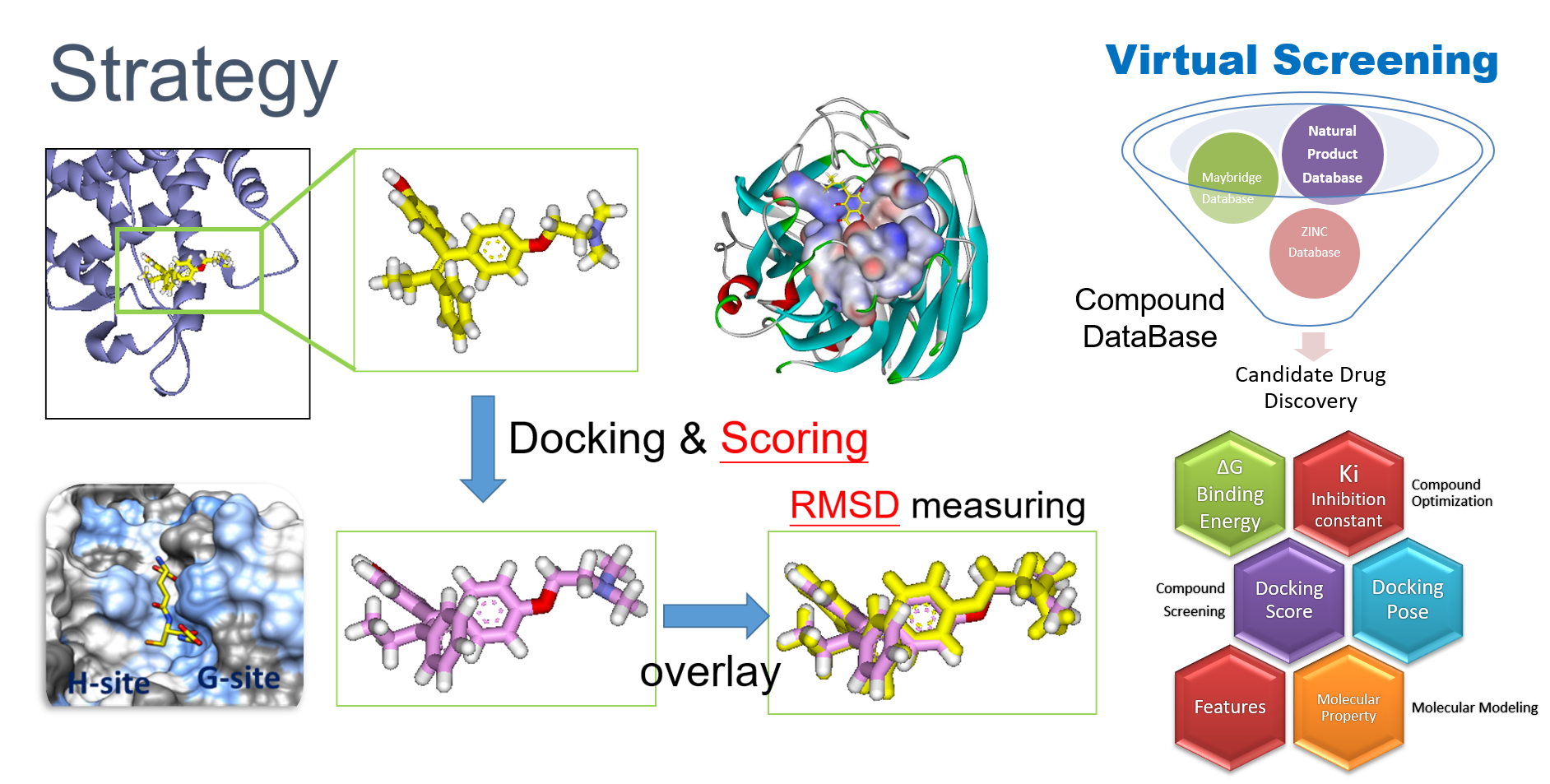
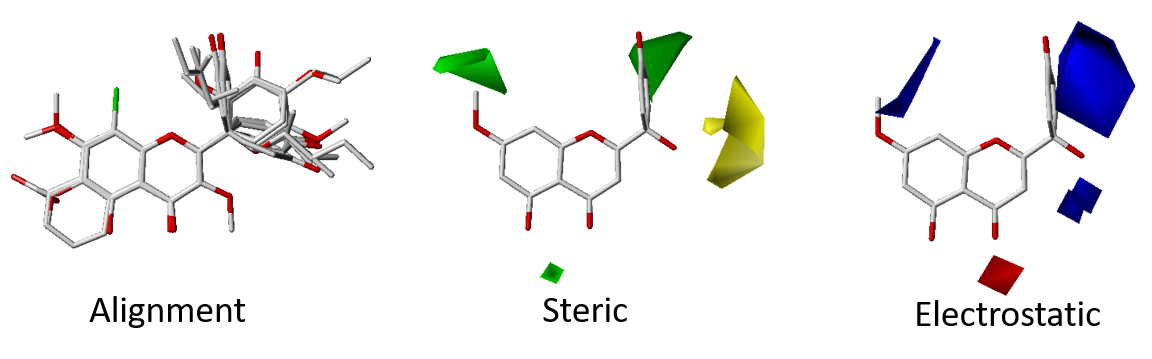
三、 定量結構活性關係(QSAR; Quantitative Structure–Activity Relationship)
本中心運用國家高速網路與計算中心提供的SYBYL軟體進行3D QSAR。3D QSAR為以小分子為基礎建立之結構與活性關係之模型。CoMFA及CoMSIA模型可以提供小分子在模擬力場上的狀態,供新藥開發研究者進一步資訊,以修飾小分子成最佳優化之潛力藥物。
服務項目
1.小分子與蛋白質對接計算模擬
2.小分子或蛋白質分子模擬分析
3.電腦虛擬藥物篩選
4.定量結構活性關係分析
服務專線
請洽本中心 (07)312-1101 #2318 / 2004 rcnpdd@kmu.edu.tw
或洽吳永昌主任 (07)312-1101 #5347 yachwu@kmu.edu.tw

